spVelo – új távlatok a génkifejeződés mérésében
A Penn State és a Yale Egyetem munkatársai által kifejlesztett spVelo nevű módszer egyszerre vizsgálja a sejten belüli térbeli információkat és azokat az adatokat, amelyeket különböző időpontokban, eltérő laboratóriumi feltételek mellett dolgoztak fel. Különösen fontos kiemelni, hogy az spVelo az ún. RNS-áramlás (RNA velocity) kiszámítására szolgál – ez azt mutatja meg, milyen irányban és milyen ütemben változik a génkifejeződés egyetlen sejten belül. Ezáltal a kutatók képesek feltérképezni, hogy egy sejt milyen típusúvá válhat a jövőben, sőt akár azt is, mely gének kapcsolódnak be vagy ki éppen abban a pillanatban.
Az RNS útja: átíródás, vágás, fehérjeszintézis
A génkifejeződés folyamata során először az örökítőanyag, vagyis a DNS kódja íródik át hírvivő RNS-re (mRNS), amelyet később fehérjékké fordít le a sejt. Az mRNS azonban szerkesztésen megy keresztül: a nem kódoló, úgynevezett intronokat kivágja a sejt, és csak a hasznos exonokat illeszti össze. Az scRNA-seq (egysejt RNS-szekvenálás) nevű technikával pontosan mérhető, hogy hány darab feldolgozott (spliced), illetve még feldolgozatlan (unspliced) RNS-molekula található egy sejten belül. Ezzel a módszerrel megállapítható, hogy éppen aktiválódik vagy visszaszorul egy adott gén működése.
Adatintegráció és térbeli információk: az igazi újdonság
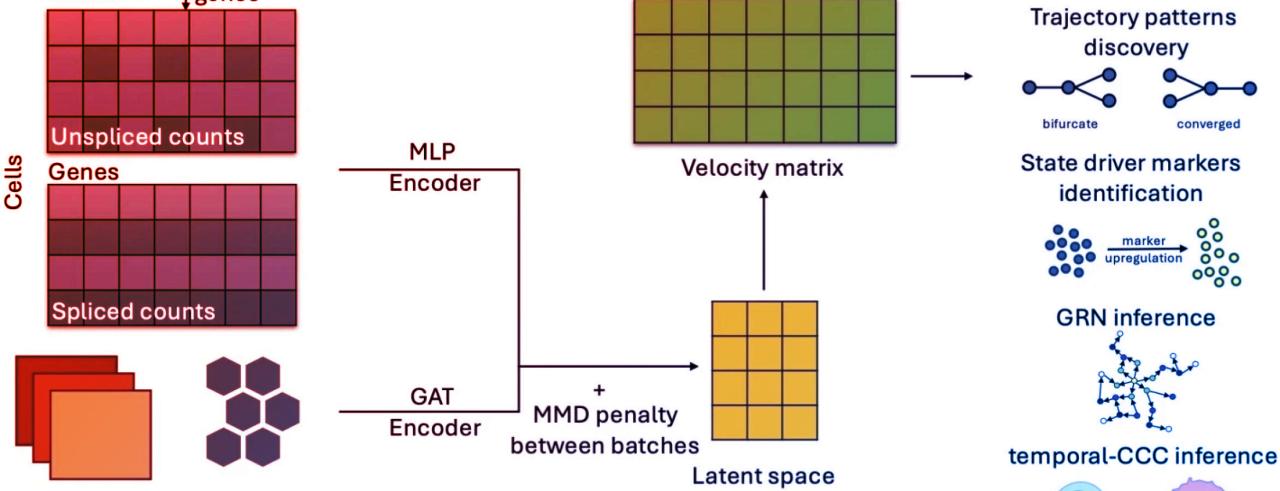
A sejtek RNS-eit gyakran eltérő időpontokban, különböző kutatócsoportok dolgozzák fel, így a laboratóriumi körülmények kis eltérései befolyásolhatják a mért eredményeket. Ezzel szemben az spVelo képes egyszerre több, külön adagban (batch) készült adatsoron is dolgozni, kiküszöbölve az eltérésekből adódó torzításokat, és így sokkal komplexebb, részletesebb képet ad a sejtek génkifejeződéséről. Másik jelentős újítása, hogy a térbeli információkat is beépíti: így már az is megtudható, hogy egy sejt pontosan hol helyezkedik el a szöveten belül, és ez hogyan befolyásolja a génkifejeződését.
MI-hálózatok: az adatok új urai
Az spVelo a mesterséges intelligencia (MI) két speciális hálózatára támaszkodik: az egyik egy variációs autoencoder, amely modellezi a génkifejeződést, a másik pedig egy grafikus figyelmi hálózat (Graph Attention Network), amely lehetővé teszi mind a térbeli, mind a batch-információk integrálását. Emellett alkalmaz egy maximum mean discrepancy nevű büntetőtagot is, amely tovább növeli az összehasonlíthatóságot több adatkészlet között. Így az MI segítségével soha nem látott összetettségű sejtfejlődési utakat tudunk leírni, előre jelezve, hogy egy sejt milyen irányba specializálódhat.
A sejtsorsdöntések jelentősége
Az új módszert ráktípusokon, például a szájüregi laphámrák sejtjein, valamint modellezett hasnyálmirigy-adatokon is tesztelték; minden esetben legalább ugyanolyan jól, vagy jobban teljesített, mint a korábbi megoldások. A rendszer rámutatott, hogy a sejteknél különböző, összetett fejlődési utak is léteznek, és akár előre lehet jelezni, milyen sejttípus vagy altípus jön létre. Egyúttal becslést is ad a számítások bizonyosságáról: egyes sejteknél biztos az irány, míg másoknál többféle lehetőséget is feltételez.
Kutatás és jövő: új lehetőségek előtt
A spVelo új dimenziót nyithat a génszabályozó hálózatok vizsgálatában is: például kimutatható vele, hogy egy gén hiánya milyen hatással van a sejt jövőjére. Ráadásul, mivel az RNS-áramlás minden egyes pillanatban információt ad, időbeli változásait követve azt is megérthetjük, hogyan kommunikálnak egymással a sejtek, és milyen gyorsan.
Ez a robusztus, megbízható MI-alapú módszer lehetővé teszi több adatsor összeillesztését és a térbeli mintázatok feltérképezését, utat nyitva a sejtbiológiai kutatások új korszakához.